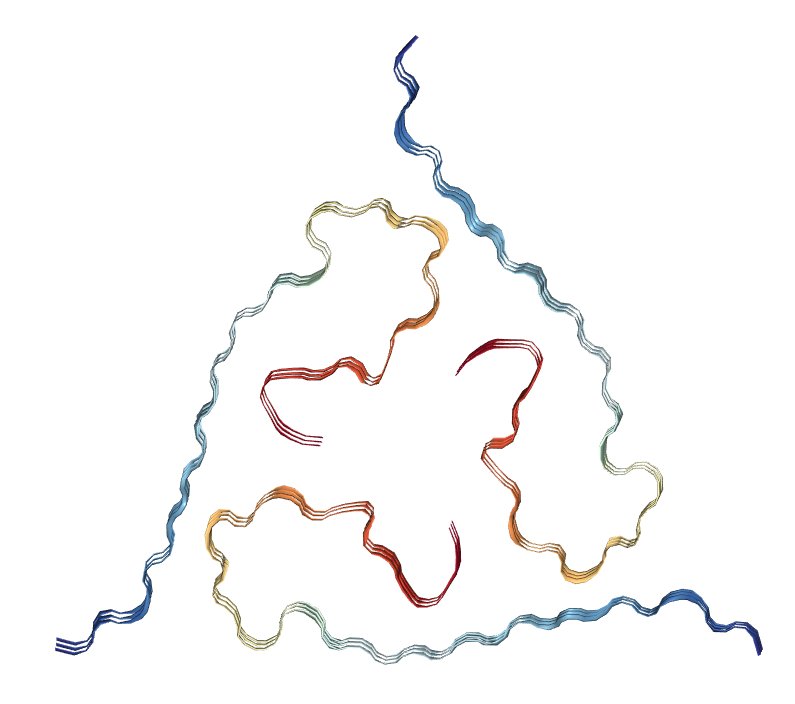
 Modello di una fibrilla amiloide-beta dell'Alzheimer. Con modelli matematici e chimici i ricercatori hanno rilevato un debole tipo di attrazione elettrica che aiuta a tenere insieme i fogli beta nelle proteine, inclusa l'amiloide-beta dell'Alzheimer. (Fonte: Lu et al.) NGL viewer e RCSB Protein Data Bank
Modello di una fibrilla amiloide-beta dell'Alzheimer. Con modelli matematici e chimici i ricercatori hanno rilevato un debole tipo di attrazione elettrica che aiuta a tenere insieme i fogli beta nelle proteine, inclusa l'amiloide-beta dell'Alzheimer. (Fonte: Lu et al.) NGL viewer e RCSB Protein Data Bank
Le interazioni chimiche che danno forma alle proteine possono essere più deboli e più numerose di quanto si sapeva finora. Queste connessioni deboli danno ai ricercatori un modo nuovo di comprendere le proteine che causano malattie e li aiutano a ottenere informazioni sui fondamenti della chimica.
Dei chimici dell'Università di Tokyo hanno modellato i mattoni della struttura proteica che causa l'Alzheimer, i fogli di amiloide-beta. I loro calcoli hanno rivelato che alcuni atomi troppo distanti dal legame si trovavano ancora nella 'zona di elettroni' di ognuno di loro.
"Questo è molto strano. È fuori del senso comune della chimica organica", ha detto il professor Tomohiko Ohwada.
Gli atomi - i mattoni della vita rappresentati nella tavola periodica degli elementi - si collegano insieme per costruire molecole, condividendo o rubando elettroni da altri atomi. Quegli elettroni volano in giro a super velocità in una nuvola di elettroni.
Quello che si sa tradizionalmente dei legami chimici è che, nelle grandi molecole, gli atomi devono essere fianco a fianco per condividere gli elettroni.
Il team di ricerca di Ohwada ha calcolato che gli atomi che sono molto distanti non condividono gli elettroni, ma le loro nuvole elettroniche possono ancora influenzarsi a vicenda. E chiamano questa influenza through-space bond path interactions (interazioni del percorso di legame nello spazio).
Queste interazioni sono estremamente deboli, ma abbastanza comuni da aggiungersi a un'influenza potenzialmente significativa sulla struttura generale delle grandi molecole. "Abbiamo studiato la struttura dell'amiloide-beta perché tutti sanno che può causare malattie, ma nessuno sa davvero come si sviluppa questa struttura problematica", ha detto Ohwada.
L'accumulo anomalo di fogli di amiloide-beta può causare il morbo di Alzheimer o dei tumori.
La struttura del foglio beta si riferisce a lunghe catene di proteine piegate a intervalli regolari e impilate una sopra l'altra in un foglio piatto. Le 'interazioni del percorso di legame nello spazio' all'interno e tra le catene proteiche probabilmente stabilizzano la struttura e possono aiutarla a raggrupparsi in placche che causano malattie.
Riconoscere la posizione e la natura delle 'interazioni del percorso di legame nello spazio' può aiutare i ricercatori a prevedere la vera struttura e il comportamento di una molecola solo sulla base della sua sequenza chimica.
Finora il team di ricerca ha studiato solo le 'interazioni del percorso di legame nello spazio' nelle porzioni idrorepellenti dei mini fogli di amiloide-beta costruiti sinteticamente. Hanno in programma di espandere i loro calcoli alle porzioni che attirano l'acqua e alle molecole più grandi.
"In teoria, potrebbe essere possibile costruire una molecola artificiale in grado di formare 'interazioni del percorso di legame nello spazio' con le proteine naturali, e cambiare la sua attività", ha detto Ohwada.
Fonte: University of Tokyo (> English text) - Traduzione di Franco Pellizzari.
Riferimenti: Luhan Zhai, Yuko Otani, Tomohiko Ohwada. Uncovering the Networks of Topological Neighborhoods in β-Strand and Amyloid β-Sheet Structures. Scientific Reports, 24 July 2019, DOI
Copyright: Tutti i diritti di eventuali testi o marchi citati nell'articolo sono riservati ai rispettivi proprietari.
Liberatoria: Questo articolo non propone terapie o diete; per qualsiasi modifica della propria cura o regime alimentare si consiglia di rivolgersi a un medico o dietologo. Il contenuto non rappresenta necessariamente l'opinione dell'Associazione Alzheimer onlus di Riese Pio X ma solo quella dell'autore citato come "Fonte". I siti terzi raggiungibili da eventuali collegamenti contenuti nell'articolo e/o dagli annunci pubblicitari sono completamente estranei all'Associazione, il loro accesso e uso è a discrezione dell'utente. Liberatoria completa qui.
Nota: L'articolo potrebbe riferire risultati di ricerche mediche, psicologiche, scientifiche o sportive che riflettono lo stato delle conoscenze raggiunte fino alla data della loro pubblicazione.





 Associazione Alzheimer OdV
Associazione Alzheimer OdV