Fino ad oggi, gli scienziati hanno identificato 32 mutazioni genetiche associate ad un aumento del rischio di morbo di Alzheimer, e la Brigham Young University (BYU) sta contribuendo a molte di quelle scoperte. Trovare questi collegamenti è una importante ricerca fondazionale, ma è estremamente impegnativo capire esattamente come queste varianti genetiche influenzano la biologia umana e cambiare realmente il rischio della malattia.
Un nuovo studio interdisciplinare pubblicato su Nature Scientific Reports, eseguito da docenti e studenti della BYU ha fatto proprio questo, scoprendo in laboratorio le differenze specifiche causate da una mutazione del gene legato al MA. Lo studio rappresenta un passo enorme per avvicinarsi allo sviluppo di trattamenti più efficaci per i pazienti diagnosticati con la malattia neurodegenerativa.
“Questo è iniziato come un salto nel buio”, ha detto l'autore senior Scott Weber, professore di microbiologia e biologia molecolare. “Ci sono molti ricercatori che hanno identificato i geni associati a un fenotipo, ma non hanno caratterizzato con successo il loro funzionamento a livello molecolare. È abbastanza impressionante vedere che questo progetto ha funzionato così bene”.
Lo studio ha richiesto anni per essere completato, con il primo autore e neodottorato Josue Gonzalez che ha lavorato sotto la guida sia di Weber (competente in immunologia molecolare) che del collega autore senior John “Keoni” Kauwe, esperto di genetica e MA. Gonzalez ha effettuato la maggior parte del lavoro tecnico e complesso di laboratorio, basato su anni di scienza e su studi precedenti.
La scienza (molto tecnica)
Studi hanno dimostrato che un aumento dei livelli di una certa proteina 'citochina' presenti nel cervello e nel fluido spinale è associata ad un rischio più alto di MA. Questa proteina, chiamata CCL2, è importante perché ha un ruolo nel reclutamento di cellule immunitarie ai siti di neuroinfiammazione, la risposta infiammatoria all'interno del cervello o del midollo spinale.
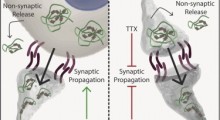
I livelli di questa proteina CCL2 nel sito di infiammazione sono alterati dal modo in cui si lega a vari recettori e cellule che attiva. Un'analisi genomica guidata da Kauwe ha identificato una mutazione genetica in uno di questi recettori delle cellule che portano ad un aumento dei livelli di CCL2 circolanti nel sangue e nel liquido cerebrospinale.
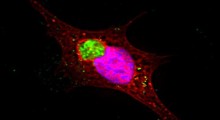
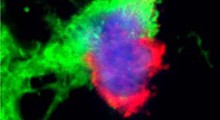
Attraverso una serie di esperimenti di laboratorio, i ricercatori della BYU hanno scoperto che la versione mutata di questo 'recettore atipico chemochine' non è in grado di legarsi altrettanto bene dei recettori normali alla proteina CCL2 del sistema immunitario associata al MA.
“Abbiamo preso le varianti dei recettori in questione e le abbiamo messe nelle cellule per vedere come si sarebbero legate alle citochine”, ha detto Weber. “Abbiamo potuto vedere che c'erano livelli più elevati di infiammazione con i recettori atipici chemochine, il che significa che non si sbarazzano così bene della citochina infiammatoria. E come sappiamo, livelli più elevati di quella citochina infiammatoria (CCL2) implicano maggiore rischio di MA“.
L'impatto (molto promettente)
Mentre solo un sottogruppo di pazienti con MA ha questa variante del recettore atipico, questo lavoro apre nuove opportunità per gli scienziati per capire come questa popolazione ha un rischio maggiore di neuroinfiammazione e di MA. E una migliore comprensione di come funziona questo recettore potrebbe portare a trattamenti efficaci che abbassano i fattori di rischio per questo gruppo.
“È stato emozionante scoprire che il recettore mutante si è comportato in modo diverso rispetto a quelli che consideriamo normali”, ha detto Gonzalez. “Questi tipi di recettori non sono stati ben studiati e la conoscenza che abbiamo di loro è limitata. Con questo progetto, abbiamo dimostrato che questi recettori possono essere un percorso per gestire lo sviluppo di malattie correlate al sistema immunitario e alla neuroinfiammazione”.
Anche i professori Perry Ridge (Dipartimento di Biologia), Eric Wilson e Julianne Grose (entrambi del Dipartimento di Microbiologia e Biologia Molecolare) della BYU hanno contribuito ad orientare lo studio attraverso strati di bioinformatica, genetica, biochimica e biologia molecolare. Kauwe ha detto che lo studio è uno dei pochi esempi di caratterizzazione biologica attenta della variante causale che rappresenta un risultato significativo di analisi genetica statistica su larga scala.
“L'abbiamo preso da grandi analisi di dati statistici e abbiamo tracciato tutto il viaggio a ritroso fino alle proprietà biochimiche specifiche della variante genetica”, ha detto Kauwe. “Abbiamo passato anni a sviluppare le basi con il nostro lavoro genetico, e ora con questo manoscritto, stiamo ponendo i primi mattoni di questo edificio”.
Fonte: Todd Hollingshead in Brigham Young University (> English text) - Traduzione di Franco Pellizzari.
Riferimenti: Josue Gonzalez Murcia, Allen Weinert, Claudia Tellez Freitas, Daniel Arens, Meganne Ferrel, Julianne Grose, Perry Ridge, Eric Wilson, John Kauwe, Scott Weber. Atypical chemokine receptor ACKR2-V41A has decreased CCL2 binding, scavenging, and activation, supporting sustained inflammation and increased Alzheimer’s disease risk, Scientific Reports, 15 May 2020, DOI
Copyright: Tutti i diritti di eventuali testi o marchi citati nell'articolo sono riservati ai rispettivi proprietari.
Liberatoria: Questo articolo non propone terapie o diete; per qualsiasi modifica della propria cura o regime alimentare si consiglia di rivolgersi a un medico o dietologo. Il contenuto non rappresenta necessariamente l'opinione dell'Associazione Alzheimer onlus di Riese Pio X ma solo quella dell'autore citato come "Fonte". I siti terzi raggiungibili da eventuali collegamenti contenuti nell'articolo e/o dagli annunci pubblicitari sono completamente estranei all'Associazione, il loro accesso e uso è a discrezione dell'utente. Liberatoria completa qui.
Nota: L'articolo potrebbe riferire risultati di ricerche mediche, psicologiche, scientifiche o sportive che riflettono lo stato delle conoscenze raggiunte fino alla data della loro pubblicazione.







 Associazione Alzheimer OdV
Associazione Alzheimer OdV