Usando i dati della sequenza dell'esoma, un team internazionale ha identificato varianti al di fuori del locus APOE che sembrano mediare il rischio di morbo di Alzheimer (MA) in individui con o senza l'allele epsilon-4 dell'APOE (APOE4), che costituisce il rischio noto per la malattia.
I membri dell'Alzheimer's Disease Sequencing Project e dell'Alzheimer's Disease Exome Sequencing-France Project hanno iniziato a scavare nei dati di sequenza dell'esoma (solo la parte di DNA trascritto nell'RNA) di oltre 16.000 casi di MA e 17.500 controlli non affetti, cercando varianti che possono contribuire al rischio di MA nel contesto dello stato epsilon- 4 dell'APOE individuale.
I risultati, pubblicati ieri online su JAMA Neurology, hanno indicato una serie di varianti che potrebbero aiutare a stratificare il rischio in individui che sembrano essere inclini al MA. Le associazioni scoperte nelle analisi "rafforzano la conoscenza e suggeriscono percorsi aggiuntivi che portano al MA", ha scritto l'autore senior Lindsay Farrer, ricercatore di genetica biomedica della Boston University, e i suoi colleghi.
Sebbene il MA ad insorgenza tardiva sia collegato all'APOE4, puntare quell'allele non sembra ostacolare la malattia neurodegenerativa, hanno osservato gli autori. Ciò ha indotto a ipotizzare che il genotipo APOE ad alto rischio "potrebbe essere un surrogato o un biomarcatore del meccanismo causale", hanno spiegato, o che l'allele epsilon-4 "potrebbe avere un ruolo in più percorsi che portano al MA o dipendere da altri fattori genetici o non genetici".
Nel tentativo di trovare nuovi contributori al MA, i ricercatori hanno messo insieme i dati di sequenza dell'esoma di 5.522 individui non legati al MA e più di 4.900 individui di controllo con cognizione normale, tutti con ascendenza bianca non ispanica. Dopo i passaggi dei controlli di qualità, sono rimasti i dati di 2.377 casi di MA e 706 controlli ancora non affetti, tutti portatori dell'allele APOE4, insieme a dati che rappresentano 3.145 individui con MA e più di 4.200 individui di controllo che non avevano la versione di APOE correlata al MA.
Il team ha tentato di convalidare i potenziali contributori di MA usando i dati di altre due coorti: dati dell'esoma di altri 1.766 individui con MA e 2.906 senza, così come 8.728 individui con MA e 9.808 controlli valutati mediante genotipizzazione basata su array (=matrice).
A seconda del tipo di analisi dell'associazione usata, i ricercatori hanno rintracciato fino a 22 varianti con potenziali legami con il rischio di MA. Dal gruppo di ricerca e da una meta-analisi eseguita in combinazione con i dati delle coorti di replica, ad esempio, hanno visto un polimorfismo a singolo nucleotide (SNP, single-nucleotide polymorphism) potenzialmente protettivo nel locus ISYNA1 per i portatori dell'allele APOE correlato al MA.
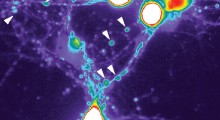
Nel gruppo senza APOE4, dall'altra parte, le analisi del team hanno evidenziato potenziali SNP associati al MA in AC099552 e in GPAA1, compresa una variante chiamata rs138412600 che sembra influenzare l'espressione di GPAA1 e un fattore repressivo di trascrizione correlato chiamato FOXG1, nel cervello.
"La bioinformatica e gli studi funzionali della variante rs138412600 in GPAA1, che era il segnale di associazione innovativo più robusto, hanno dimostrato che potrebbe anche essere associato alla funzione cognitiva globale e all'espressione di GPAAA1 nel cervello e nel suo fattore di trascrizione repressivo, FOXG1", hanno segnalato gli autori.
Nel gruppo di casi e controlli privi dell'allele APOE4 di alto rischio, i ricercatori hanno anche segnalato SNP con associazioni più incerte in, o intorno a, geni precedentemente implicati nel rischio di AD, tra cui MAPT e TREM2.
Tuttavia, gli autori hanno avvertito che i geni specifici implicati nelle loro analisi "dovrebbero essere considerati provvisori", poiché "i set di dati di replica non erano adatti per il test di replicazione, perché le varianti sentinella che contavano per le associazioni non erano presenti o non ben definite in questi campioni".
Fonte: GenomeWeb (> English text) - Traduzione di Franco Pellizzari.
Riferimenti: Yiyi Ma, Gyungah R. Jun, Xiaoling Zhang, Jaeyoon Chung, Adam C. Naj, Yuning Chen, Celine Bellenguez, Kara Hamilton-Nelson, Eden R. Martin, Brian W. Kunkle, Joshua C. Bis, Stéphanie Debette, Anita L. DeStefano, Myriam Fornage, Gaël Nicolas, Cornelia van Duijn, David A. Bennett, Philip L. De Jager, Richard Mayeux, Jonathan L Haines, Margaret A. Pericak-Vance, Sudha Seshadri, Jean-Charles Lambert, Gerard D. Schellenberg, Kathryn L. Lunetta, Lindsay A. Farrer, for the Alzheimer’s Disease Sequencing Project and Alzheimer’s Disease Exome Sequencing–France Project. Analysis of Whole-Exome Sequencing Data for Alzheimer Disease Stratified by APOE Genotype. JAMA Neurology, 10 June 2019, DOI: 10.1001/jamaneurol.2019.1456
Copyright: Tutti i diritti di eventuali testi o marchi citati nell'articolo sono riservati ai rispettivi proprietari.
Liberatoria: Questo articolo non propone terapie o diete; per qualsiasi modifica della propria cura o regime alimentare si consiglia di rivolgersi a un medico o dietologo. Il contenuto non rappresenta necessariamente l'opinione dell'Associazione Alzheimer onlus di Riese Pio X ma solo quella dell'autore citato come "Fonte". I siti terzi raggiungibili da eventuali collegamenti contenuti nell'articolo e/o dagli annunci pubblicitari sono completamente estranei all'Associazione, il loro accesso e uso è a discrezione dell'utente. Liberatoria completa qui.
Nota: L'articolo potrebbe riferire risultati di ricerche mediche, psicologiche, scientifiche o sportive che riflettono lo stato delle conoscenze raggiunte fino alla data della loro pubblicazione.




 Associazione Alzheimer OdV
Associazione Alzheimer OdV