 Una collaborazione tra scienziati dello Scripps Research Institute (TSRI) e dell'Università della California (UC) di San Diego ha sviluppato un nuovo potente sistema per studiare come le proteine e le altre molecole biologiche si formano e perdono la loro struttura naturale piegata.
Una collaborazione tra scienziati dello Scripps Research Institute (TSRI) e dell'Università della California (UC) di San Diego ha sviluppato un nuovo potente sistema per studiare come le proteine e le altre molecole biologiche si formano e perdono la loro struttura naturale piegata.
Con il nuovo sistema, i ricercatori possono forzare un campione di molecole a dispiegarsi e a ripiegarsi, aumentando e quindi facendo cadere la temperatura, così velocemente da riuscire a tracciare anche gli eventi di piegatura molecolare più veloci .
"Uno dei modi di studiare queste strutture è indurle a dispiegarsi o a piegarsi con il calore, e osservare la cinetica e le altre proprietà di quegli eventi di dispiegamento ed ripiegamento", ha detto Ashok A. Deniz, Professore Associato del TSRI. "Il nuovo sistema ci permette di fare questo in modo da superare alcune delle limitazioni principali dei metodi precedenti".
L'invenzione può essere applicata non solo allo studio di normali biomolecole, ma anche a molte di quelle anormali mal ripiegate, che sono implicate nelle malattie umane. I risultati del laboratorio di Deniz e di quello di uno stretto collaboratore, il biofisico Alex Groisman della UC San Diego, sono pubblicati in Nature Communications questa settimana.
Troppo piccolo, troppo veloce
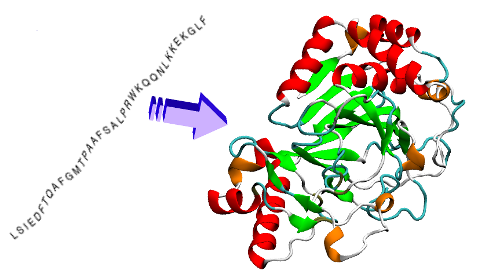
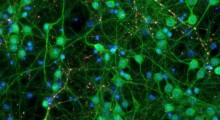
Studiare come si piegano le proteine, il DNA e le altre molecole biologiche è una delle aree più difficili della biologia, in particolare perché queste piccole molecole - più piccole delle lunghezze d'onda della luce visibile - possono piegarsi o dispiegarsi a intervalli di pochi milionesimi di secondo (microsecondi).
I ricercatori in questo campo hanno apportato alcune potenti innovazioni più o meno negli ultimi dieci anni, compreso l'uso di fenomeni ottici quantistici esotici per monitorare i movimenti molecolari rapidi. Ma mancavano tecniche per perturbare le molecole piegate con improvvisi cambiamenti di temperatura. "La dimensione dei salti di temperatura era limitata a circa 15 gradi Celsius, e mancava un modo per raffreddare i campioni rapidamente alla temperatura ambiente", ha dichiarato Deniz.
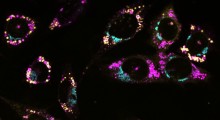
Nel nuovo studio, i laboratori di Deniz e Groisman si sono uniti per sviluppare un dispositivo che superasse entrambe le limitazioni. Il loro dispositivo microfluidico, come altri che sono stati sviluppati in questo campo, è un piccolo blocco di silicone dotato di piccoli canali attraverso i quali può fluire una soluzione contenente biomolecole. In questo caso, il canale di osservazione è profondo meno di un milionesimo di metro, è foderato di zaffiro per condurre il calore rapidamente, e contiene anche una doratura sottile per assorbire efficacemente la potenza di un fascio laser a infrarossi e convertirlo in calore.
La combinazione tra il piccolo volume di riscaldamento e la doratura per l'assorbimento dell'energia implica che il laser può riscaldare un campione di molecole molto rapidamente, con un salto di temperatura superiore a 50 gradi Celsius in meno di un microsecondo. Il substrato di zaffiro termoconduttore a sua volta permette al campione di raffreddarsi di diverse decine di gradi, di nuovo entro circa un microsecondo, non appena il laser è spento.
Dimostrazioni innovative
Come semplice prova dell'utilità del nuovo dispositivo, i ricercatori l'hanno usato per riscaldare e raffreddare la soluzione di un breve filamento di DNA che forma naturalmente un anello a forma di forcina (hairpin-shape). Le «Hairpin DNA» sono strutture dinamiche con un ruolo chiave nella replicazione cellulare e in altri importanti processi biologici.
L'esperimento, che comprendeva tecniche di fluorescenza per rilevare stati piegati o non piegati del filamento di DNA, ha rivelato che il piegamento a forcina può formarsi in pochi microsecondi. "Non avremmo potuto osservare una tale rapida piegatura con i sistemi precedenti di salto di temperatura, che in genere ci mettono migliaia di microsecondi per raffreddare le molecole", ha detto Priya R. Banerjee, socio di ricerca nel laboratorio di Deniz, e primo autore della relazione con Mark E. Polinkovsky del laboratorio di Groisman, e Yann Gambin, anch'egli membro del laboratorio di Deniz al tempo dello studio.
Inoltre, gli scienziati hanno usato la nuova configurazione per studiare una forcina più grande di DNA, insieme alla conferma del comportamento previsto in precedenza sull'effetto differenziale del cloruro di sodio (sale ordinario) sulla cinetica della piegatura e dispiegatura di forcine di dimensioni diverse.
In una dimostrazione finale sulle nuove funzionalità della loro nuova configurazione, gli scienziati sono riusciti a vedere che il sistema molecolare si comporta come un filtro a basso passaggio, simile in qualche modo a quelli elettronici o ai circuiti cellulari. Il team ha usato la capacità di commutare il laser riscaldante, accendendolo e spegnendolo rapidamente, in combinazione con le caratteristiche di riscaldamento e di raffreddamento rapido del nuovo sistema, consentendo nuovi tipi di studi sugli scenari della piegatura molecolare.
Essenzialmente, i ricercatori possono ora sottoporre una soluzione di molecole ad una serie continua di cicli di riscaldamento e raffreddamento, alterando a volontà la frequenza e la temperatura di questa gamma di oscillazione riscaldamento/raffreddamento e osservando la dinamica del piegamento / ri-piegamento delle molecole. "Prevediamo che questo tipo di esperimento ci permetterà di rilevare le caratteristiche più complesse o nascoste dei paesaggi strutturali biomolecolari che non sono mai stati visti prima", ha detto Deniz.
Deniz ed i suoi colleghi hanno ora intenzione di usare il loro nuovo sistema per studiare la dinamica di piegatura di altre molecole, comprese le proteine soggette alla mal-ripiegatura che possono causare malattie umane comuni come l'Alzheimer, il Parkinson e l'Huntington.
*******
Oltre a Deniz, Groisman, Banerjee, Gambin e Polinkovsky, ha collaborato allo studio Michael J. Erickstad della UC San Diego. Il supporto per la ricerca è stato dato dalla National Science Foundation.
Fonte: The Scripps Research Institute (> English text) - Traduzione di Franco Pellizzari.
Riferimenti: Mark E. Polinkovsky, Yann Gambin, Priya R. Banerjee, Michael J. Erickstad, Alex Groisman, Ashok A. Deniz. Ultrafast cooling reveals microsecond-scale biomolecular dynamics. Nature Communications, 2014; 5: 5737 DOI: 10.1038/ncomms6737
Copyright: Tutti i diritti di eventuali testi o marchi citati nell'articolo sono riservati ai rispettivi proprietari.
Liberatoria: Questo articolo non propone terapie o diete; per qualsiasi modifica della propria cura o regime alimentare si consiglia di rivolgersi a un medico o dietologo. Il contenuto non dipende da, nè impegna l'Associazione Alzheimer onlus di Riese Pio X. I siti terzi raggiungibili da eventuali links contenuti nell'articolo e/o dagli annunci pubblicitari sono completamente estranei all'Associazione, il loro accesso e uso è a discrezione dell'utente. Liberatoria completa qui.
Nota: L'articolo potrebbe riferire risultati di ricerche mediche, psicologiche, scientifiche o sportive che riflettono lo stato delle conoscenze raggiunte fino alla data della loro pubblicazione.
| Sostieni l'Associazione; una donazione, anche minima, ci aiuterà ad assistere malati e famiglie e continuare ad informarti. Clicca qui a destra: |





 Associazione Alzheimer OdV
Associazione Alzheimer OdV